Gibson cloning 이란?
제한 효소 없이 overlapping ends 를 이용하여 cloning 하는 방법.
Strand chew back→ expossing single strand → overlapping ends annealing → 3' extension → Ligation
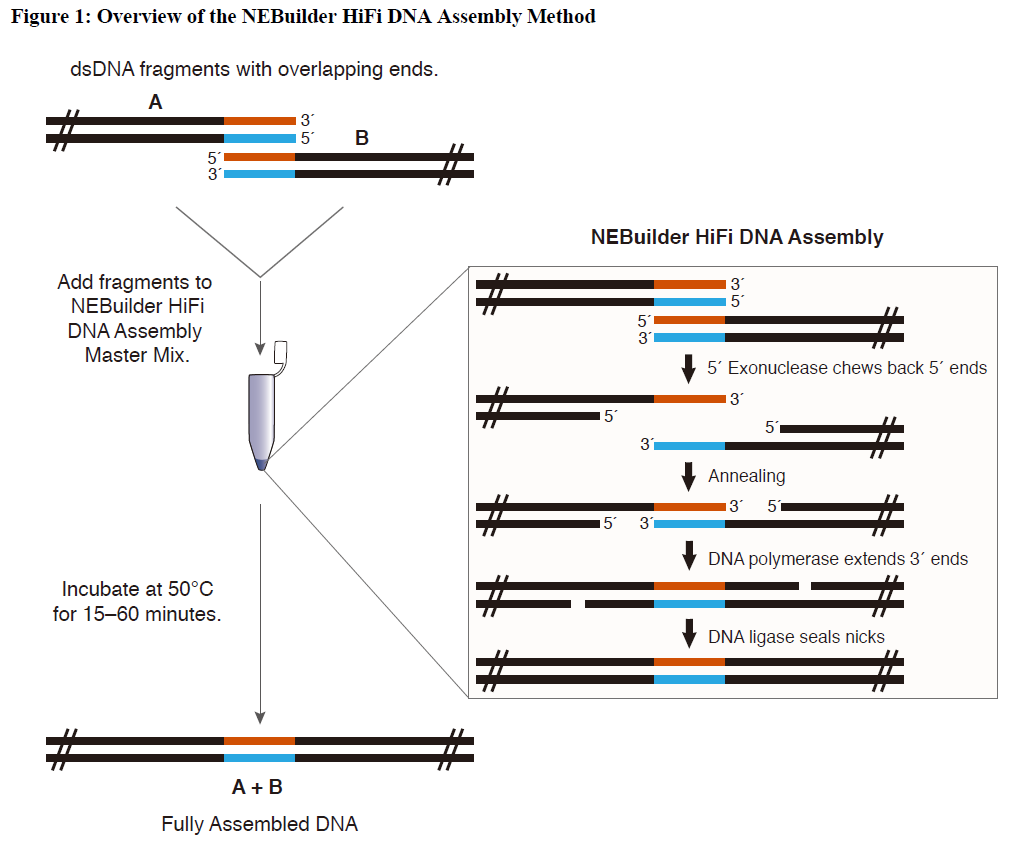
Overlapping ends 만 잘 디자인 하면 여러개의 product로 한번에 insertion 가능하다는 장점이 있다.
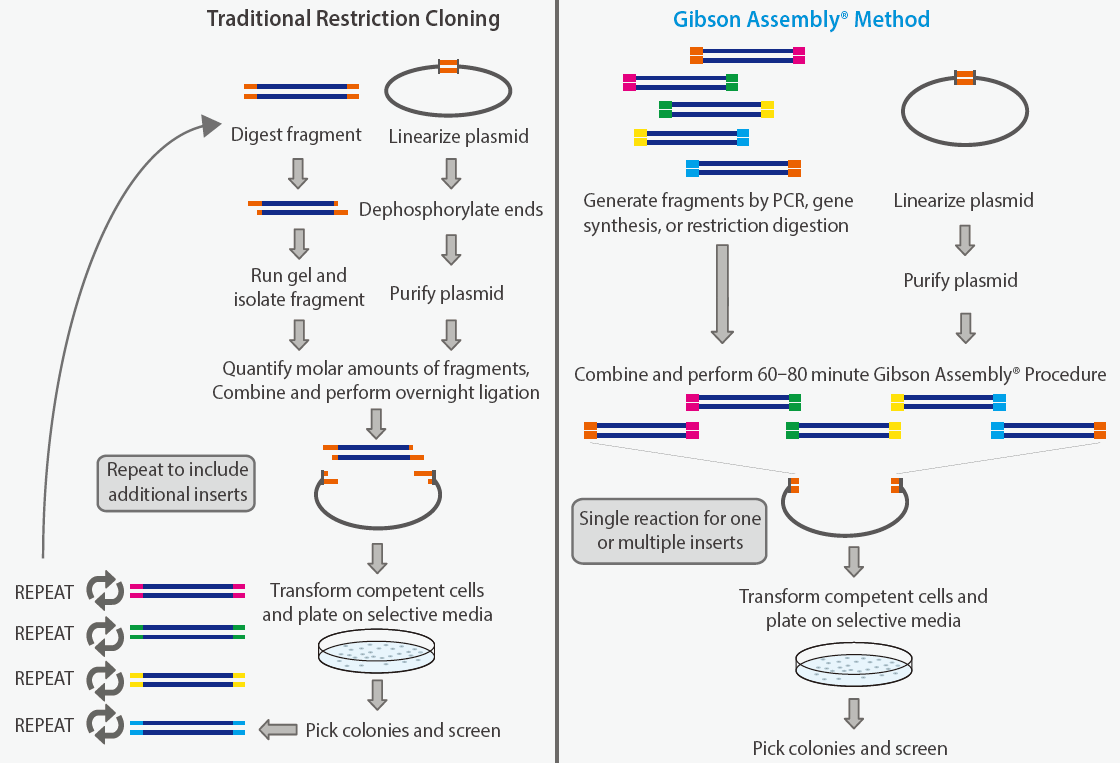
Gibson cloning을 위해서는 overlapping ends 를 가진 linearize vector 와 insert 가 필요하다.
1. PCR
이 방법은 vector 와 insert 모두 PCR 하는 방법이다.
Overlapping region 만 잘 설정하면 된다.

2. Restriction enzyme digested vector
이 방법은 기존에 가지고 있는 vector 를 제한 효소로 잘라서 준비하는 방법이다.
하나의 제한 효소보다는 두가지 제한효소로 자르는 것이 좋다. (uncut background를 줄이기 위하여)
(하나의 제한 효소로 자르더라도 ligation system이 아니므로 CIP 처리는 하지 않아도 된다.)
PCR을 한번만 해도 된다는 장점이 있지만 Restriction enzyme site를 잘 고려하여 primer를 디자인 해야 한다.
Frame 을 신경써서 맞추는 것이 좋다! (특히 C-terminal tagging 이 필요한 경우 주의할 것!)

연습
primer design for mClover3 and KpnI-cut-pcDNA3 assembly

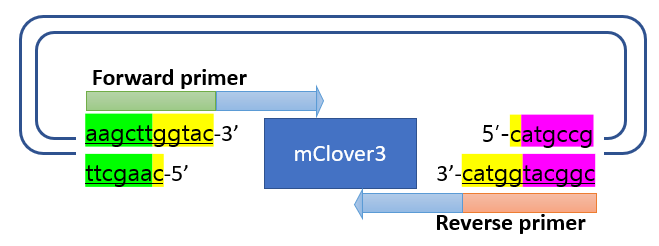
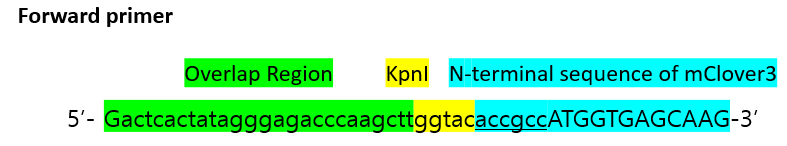
Forward primer
5' chew back 이므로 3' end 까지 맞추면 된다.
KpnI site가 사라지는 시스템
시작 부분이므로 kozak sequence 과 start codon이 필요하다.
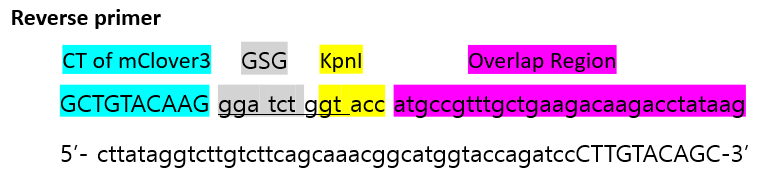
Reverse primer
뒷부분에 다른 gene이 발현되도록 하기 위해서 mClover3 뒤에 no stop 으로 디자인하였다.
GSG linker를 추가하고 3' end 에 맞춤.
overlapping ends는 ggtac 만 맞추면 되지만 frame 을 맞추기 위하여 g를 추가함으로써 KpnI이 남아있게 되었다.
https://www.biocat.com/bc/files/Gibson_Guide_V2_101417_web_version_8.5_x_11_FINAL.pdf
'BT > Lab' 카테고리의 다른 글
| 1. PCR이란? 구성요소, 원리, 과정, 조성, protocol, 레시피 (1) | 2024.04.03 |
|---|---|
| competent cell 정의, 종류 (0) | 2024.04.02 |
| 유전자 재조합 DNA cloning (1) Gateway cloning - Entry clone 만들기 (0) | 2023.03.29 |
| NCBI에서 계통수 phylogenetic tree 만들기 / Orthologs / COBALT (1) | 2021.05.27 |
| ClustalW Clustal Omega 사용방법 (0) | 2021.05.26 |




댓글