CRISPR - Cas9 system을 이용한 Knock out cell line 만들기 (1) sgRNA design
이 포스팅은 CRIPSR Cas system 을 이용하여 knock out cell line 만들기에 대한 포스팅이다. 이론에 관한 것은 잘 정리된 것이 너무 많기 때문에 PASS하고 cell line 만들 때 고려해야 하는 점등을 위주로 포스
arumyworld.tistory.com
앞의 포스팅에서 sgRNA design에 대해서 다루었다.
sgRNA cloning이 끝나면 원하는 세포에 넣어주면 끝인데
하지만 그것은 쉽지 않지... 세세한 과정을 알아보도록 하겠다.
sgRNA cloning을 하면서 cell line에 antibotics test를 진행하여야 한다.
내가 사용하는 cell line에 내가 사용하는 sgRNA를 selection 할 수 있는 antibotics를 확인하고 어떤 농도에서 가장 세포가 잘 죽는지 확인해야 한다.
사람들이 주로 사용하는 세포들과 항생제는 그 농도에 관한 정보가 잘 나와있어서 그것을 바탕으로 진행하면 된다.
농도별로 treat한 다음 보통 1일 ~3일 이내로 잘 죽는지 확인해야 한다.
sgRNA를 넣은 뒤에 확인한 농도의 antibotics를 처리하면 sgRNA가 들어있는 세포만 selection이 가능하다.
selection 진행 후 7일 정도 지나면 sigle cell colony 형태가 만들어지는데 이 때 그 colony를 picking해주면 된다. 또는 96 well plate 등을 사용하여 한 well 에 세포가 1개가 들어가도록 seeding 하여 single cell colony 를 얻을 수 있다.
single cell colony picking이 끝나고 세포가 어느 정도 자라게 되면 sequencing을 통하여 gene editing 이 어떻게 되었는지 확인할 수 있다. 이를 위하여 다음으로 진행해야 할 일은 sequencing을 위한 genomic DNA PCR primer 주문이다.
antibotics를 이용하여 selection이 끝나게 되면 그 세포가 어떤 genomic DNA pattern을 가지고 있는지 확인하여야 한다. selection 된 세포에서 genomic DNA를 prep하면 이 때 주문한 PCR primer를 이용하여 PCR을 진행하고 그 PCR product를 sequencing을 보내 정확한 sequence를 확인할 수 있다.
보통 sequencing은 target sequence 200-300bp 앞 뒤에 있는 sequence를 선택해주면되고 PCR product 예상 길이는 400-600bp가 된다. 그런데 여기서 조심해야 할 점은 우리는 genomic DNA sequence를 확인해야 하므로 CDS가 아닌 intron을 포함한 전체 genome sequence를 본다는 점이다.
UCSC Genome Browser Home
genome.ucsc.edu
genomic DNA sequence를 확인하는 많은 방법이 있겠지만 이 site를 이용하면 exon 위치를 다 알려주어 좋은 듯 하다. (사실 다른 건 안 써봄.)

Site에 들어가서 BLAT를 누르면

이런 화면이 나오고 내가 target 하는 gene sequence를 넣어주면 genome sequence가 나온다.

이렇게 list가 나오면 details 을 누르면 된다.
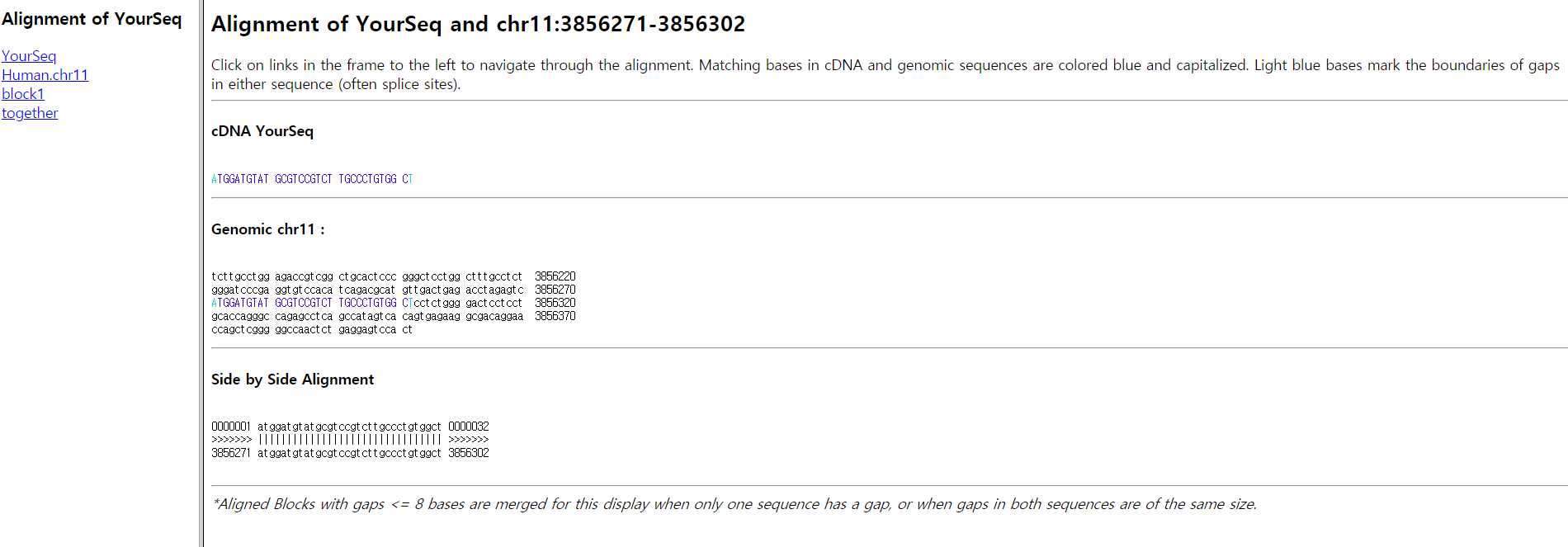
지금 예시로 나온 이미지는 sequence를 너무 짧게 넣어서 이렇게 나오지만 내가 target 하는 gene 전체를 넣으면 전체 genomic sequence가 나온다. 거기서 내가 target 하는 sequence 앞 200-300bp 앞에 있는 sequence를 이용하여 primer를 주문하면 된다. 이 sequnce가 exon에 있으면 나중에 cDNA level에서 확인할 때 다시 primer를 주문하지 않아서 되도 좋은데 exon과 exon 사이 intron의 길이가 너무 길면 그럴 수 없다.
sequencing은 세포의 karyotype에 따라서 난이도가 달라진다. 내가 edit 하고자 하는 gene이 어떤 choromosome에 위치하고 있는지 알아야하며 내가 사용하는 세포가 몇개의 chromosome을 가지고 있는지 알아야 한다.
예를 들어 내가 target 하는 gene이 chromosome 11번에 있는데 내가 사용하는 cell line이 chromosome 11번이 4개가 있을 수 있다. -> 최대 4가지 pattern 이 나올 수 있다.

특히 cancer cell line은 쉽지 않다.
HAP1 cell 같은 경우에는 chromosome이 하나씩 있기 때문에 knock-out 할 때 많이 사용된다.
그 pattern 은 sequencing 결과에서 추론해야 한다. 겹쳐있는 sequencing peak를 보고 어디서 어디까지 deletion 또는 insertion 되었는지 확인해야 한다.
이는 쉽지는 않은 과정인데 이를 좀 더 확실하게 하기 위해서는 sequencing을 위해 얻은 PCR product를 entry vector 같은 cloning plasmid에 ligation하여 (뭐든 상관 없음, TOPO PCR cloning 매우 좋음) transformation 되어 나온 여러 colony의 sequencing을 확인하면 좋다.
마지막으로 RNA level에서 sequencing을 확인하거나 (gDNA sequencing 확인 방법과 같으나 CDS를 기준으로 target site 200-300bp 앞뒤로 primer를 design 하면 된다.) Western Blot 등 protein level 에서 knock-out 확인이 가능하다.
antibody 퀄리티가 좋다면 일단 Western Blot 으로 protein level에서 knock-out 을 확인하고 그 다음 어떤 pattern을 가지고 있는지 확인하면 좋다.
순서대로 정리하자면
1. antibotics test
2. sgRNA infection or transfection
3. antibotics selection
4. single cell colony picking
5. (Western Blot)
6. genomic DNA prep/ RNA->cDNA prep
7. target site PCR
8. sequencing
의 과정으로 knock-out cell line을 만들 수 있다.
'BT > Lab' 카테고리의 다른 글
| NCBI에서 계통수 phylogenetic tree 만들기 / Orthologs / COBALT (1) | 2021.05.27 |
|---|---|
| ClustalW Clustal Omega 사용방법 (0) | 2021.05.26 |
| Western Blot (1) SDS-PAGE (0) | 2021.05.22 |
| CRISPR - Cas9 system / Knock out cell line 만들기 (1) sgRNA design (2) | 2021.02.14 |
| 코로나19/COVID19 [SARS-CoV-2] (0) | 2020.08.23 |




댓글